2. 成都诺恩生物科技有限公司, 成都 610041;
3. 中国食品药品检定研究院, 北京 100050
2. Chengdu Nuoen Biotechnologies, LTD., Chengdu 610041, China;
3. National Institutes for Food and Drug Control, Beijing 100050, China
微小核糖核酸是一类由19~22个核苷酸组成的非编码RNA,主要参与调节各种基因表达,如直接结合到mRNA的抑制或启动子区域将抑制或者促进转录基因的翻译。由于具有早期和精确诊断的应用前景,检测人体体液(包括血液、唾液、尿液和房水)中的miRNA早已受到关注。疾病相关miRNA的探索筛选工作也在不同的疾病领域得到广泛应用。miRNA被脂质或脂蛋白复合物包裹以一种相对稳定的形式进行体内循环[1-3],因而可以作为稳定的生物标志物被检测。美国FDA科学家Shi Q在2013年撰文深入论述了血液microRNA,尤其是miRNA -122作为肝损害生物学指标,具有显著优于传统标志物(ALT)的临床应用前景,但也存在无标准化的定量检测方法的问题[4]。美国FDA在2015年8月20日发布了有关生物标志物的报告[5],认定miRNA-122作为临床亟需(Critical Need)的急性肝毒性安全生物标志物(非临床和临床),可应用于药物肝脏毒性的证明性研究、临床剂量递增的安全性研究及药物诱发的肝损伤3种情况。
同时,美国FDA科学家Shi Q再次撰文论述了miRNA作为临床生物标志物的发展受制于缺少标准化定量检测方法,并引用了一种免提取PCR-荧光毛细管电泳法技术,来阐述如何建立定量检测方法,并认为这是一种创新的检测方法[6]。
以往的工作中,利用一种免提取PCR-荧光毛细管电泳法技术,也称作miRNA来源的片段长度多态性技术(miRFLP),通过免提取RNA技术可实现微量血浆和房水样本(低至1 μL)的定量检测[7-8]。本文基于miRNA方法对微小核糖核酸122(miRNA-122)定量检测的定量下限、标准曲线、回收率、精密度进行评估,并应用该技术对临床肿瘤化疗患者的血浆样本进行了检测和验证。
1 材料与方法 1.1 样本本研究根据《赫尔辛基宣言》和《议定书》进行。四川省肿瘤医院伦理审查委员会批准。使用EDTA-NA2采血管抽取四川省肿瘤医院接受化疗治疗的患者5 mL全血,采用800 g离心力离心10 min,取上层淡黄色液体转移至1.5 mL离心管并立即置于-20 ℃保存。总共收集43例肿瘤化疗患者血样。企业校准品、参考品由成都诺恩基因科技有限公司提供。
1.2 仪器与试剂仪器:Analytikjena PowerCycler pcr仪,VeritiTM PCR仪和ABI 3730 XL测序仪。
主要材料:Triton X-114(Sigma-Aldrich),Potassium chloride(Sigma-Aldrich),RT buffer(Takara),Magnesium sulphate(Sigma-Aldrich),dNTP(Sigma-Aldrich),MMLV reverse transcriptase(RT,Takara),RNase Inhibitor(RI,NewEngland Biolabs),PCR buffer(Sigma-Aldrich),JumpStart Taq DNAse(Sigma-Aldrich),MyOne Streptavidin C1(Life technologies),Ribonuclease A(RNase A,Takara),RNA序列、cDNA引物、PCR引物和生物素标记欧米伽引物(南京金斯瑞生物科技有限公司产品)。
1.3 基于miRNA的微小核糖核酸122(miRNA -122)定量检测技术取5 μ L血浆样本加入3 5 μ L样本释放剂(Triton X-114、氯化钾、逆转录酶缓冲液、硫酸镁、无核酸酶水),4 ℃ 10分钟,75 ℃ 3分钟,充分裂解包裹miRNA的外泌体,取8 μL裂解好的血浆分别加入1 μL内对照、2 μL miRNA欧米伽探针和2 μL内对照欧米伽探针,55 ℃ 5分钟,1个循环;55 ℃ 1分钟、20 ℃ 1分钟,10个循环进行杂交。交后加入4 μL逆转录反应液(dNTP、逆转录酶、鼠源RNA酶抑制剂)在37 ℃ 5分钟,1个循环;55 ℃ 15秒、4 ℃ 1分钟、20 ℃ 1分钟、37 ℃ 30秒,40个循环;85 ℃ 5分钟,1个循环条件下进行逆转录反应。逆转录反应后加入60 μL cDNA延伸反应液[miR-122cDNA延伸引物、2×PCR buffer、dNTP、内对照cDNA延伸引物、TaqDNA聚合酶、硫酸镁、核糖核酸酶(牛胰)、无核酸酶水]在95 ℃ 2分钟、60 ℃ 10分钟,1个循环;72 ℃ 1分钟、37 ℃ 3分钟,20个循环;72 ℃ 5分钟、95 ℃ 1分钟,1个循环条件下进行cDNA延长反应。反应后,加入磁珠(C1)进行纯化,用清洗液和无核酸酶水清洗,清洗后,进行PCR扩增。加入30 μL PCR反应液(PCR扩增正向引物、PCR扩增反向引物、硫酸镁、2×PCR buffer、dNTP),在95℃ 2分钟、55℃ 10分钟,1个循环;37℃ 3分钟、55℃ 1分钟,10个循环;72℃ 1分钟,1个循环;95℃ 30秒、68℃ 3分钟、72℃ 30秒,45个循环;72℃ 10分钟,1个循环,60℃ 4小时,1个循环条件下,所有PCR扩增模板进行竞争性同步扩增,完成miRFLP的反应步骤。将得到的PCR产物用10×TE(pH 7.0)进行1:100的稀释,采用荧光毛细管电泳ABI3730检测。
1.4 性能评估技术性能评估实验设计参考《美国临床实验室标准化协会批准指南》(CLSI-EP)[9-11]、《生物样品定量分析方法验证指导原则》 [12]和美国Roche公司的关于COBAS®6800/8800系统对HIV-1核酸定量检测的性能评估[13]。
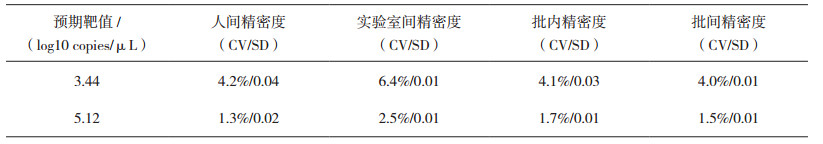
1.4.1 精密度在相同条件下(方法、试剂、仪器、参考品、实验室、操作员)对高、低两个浓度(3.44、5.12 Log10 copies/μL)水平的样本进行检测,每个样本每日2个批次,每个批次测8次,共检测5日,得到80个数据。从每个样本10个批次中每批的8次变异系数算出批内精密度。从每个样本的80个结果计算出批间精密度。不同实验员在相同条件下(方法、试剂、仪器、参考品、实验室)对高、低两个浓度水平的样本进行检测,每个样本各进行16次重复检测,计算得到人间精密度;成都诺恩基因科技有限公司在相同条件下(方法、试剂、参考品)对高、低两个浓度水平的样本进行检测,每个样本各进行16次重复检测,计算得到室间精密度。
1.4.2 检测限将样本稀释成接近于方法标准曲线低限浓度水平的3个浓度样本(1:366 copies/μL、2:183 copies/μL、3:92 copies/μL),将3个浓度样本各进行20次重复测定。
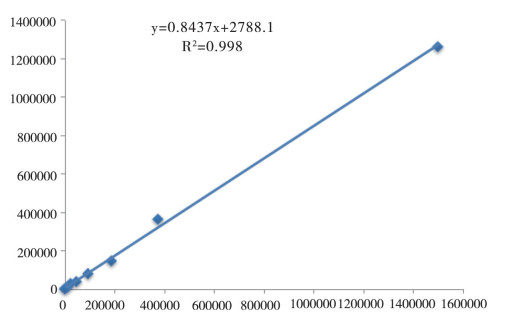
1.4.3 标准曲线准备13个不同浓度(1500000、375000、187500、93750、46875、23438、11719、5859、2930、1465、732、366、183 copies/μL)的样本进行检测。对检测结果和理论浓度进行多项式回归,得到相关系数R2。
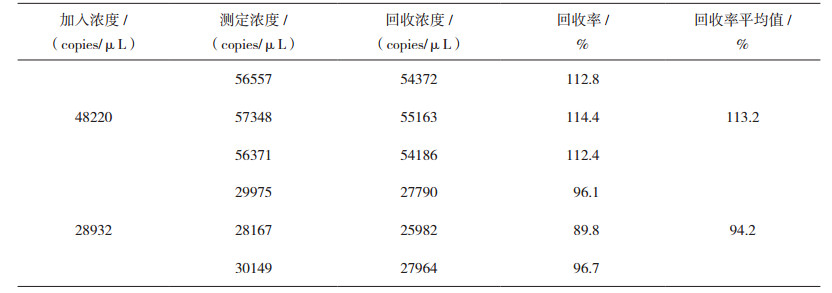
1.4.4 回收率以样本(1465 copies/μL)为标准样本,分别制备48220、28932 copies/μL的待加入样本。制备6份相同体积的待测物标准样本。每2份中加入同一浓度待加入样本,制成2个不同待加入浓度的回收样本,计算加入的待测物浓度。在2份样本中加入同样量的无被测物的样本稀释液,制成基础样本。分别对回收样本、基础样本和低浓度样本进行3次重复测定,取其均值进行计算。
1.4.5 临床样本检测单点采集化疗后24小时内43例肿瘤化疗患者的血样,并分离血浆进行miRNA-122检测,同时参照2015版《药物性肝损伤诊治指南》 [14]进行miRNA-122与现行诊断标准的ROC分析。现行诊断标准采用Roussel Uclaf因果关系评估法(RUCAM)[15-17]。
2 结果 2.1 精密度精密度结果见表 1。
|
|
表 1 精密度检测结果 |
依据技术要求,该技术可以准确地检测miRNA-122,具有很好的稳定性。
2.2 检测限结果显示,浓度为366 copies/μL时检出率是100%,浓度为183 copies/μL时检出率是95%,浓度为92 copies/μL时检出率是75%。依据技术要求,该技术定量下限为183 copies/μL。
2.3 标准曲线标准曲线结果见图 1,结合前面检测限实验结果,标准曲线为183~1500000 copies/μL。结果表明,该技术具有较宽的标准曲线,适于临床低浓度样本的检测。
|
图 1 标准曲线检测结果 |
回收率结果见表 2。
|
|
表 2 回收率实验结果 |
实验结果显示,高浓度平均回收率为113.2%,低浓度平均回收率为94.2%,比例系统误差为3.7%。依据技术要求,该技术具有很好的准确性。
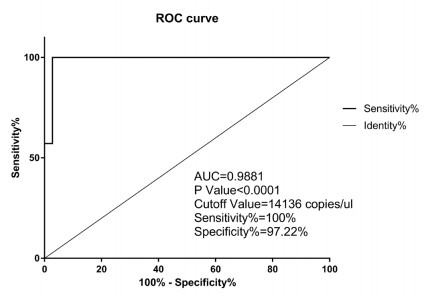
2.5 临床样本检测在现行诊断标准(ALT、R值)能够诊断的病例中,以现行诊断标准(ALT、R值)为参照,对比miRNA-122的ROC分析(见图 2)。miRNA-122与现行诊断标准(ALT、R值)具有较高的一致性,其AUC、P值、特异性及灵敏度均满足临床需求,初步确定的CutOff值设定为14136 copies/μL,该值与欧盟2016年9月30日公布的PSHT HV健康志愿组(欧洲人群)miRNA-122的13356 copies/μL(95%)基本一致[18]。
|
图 2 miRNA-122ROC分析图 |
血液中miRNA的检测技术平台主要有Northern blotting法[19-21]、微阵列芯片法[22-24]、RTqPCR[25-27]。但Northern blotting法灵敏度低、耗时长;微阵列芯片法稳定性和重复性差,无法实现定量检测;RT-qPCR是最为广泛应用的检测技术,但该方法需要提取总RNA后再进行检测,样本需求量大,同时还需要找到内源性参考物来估量提取过程靶标的损失,限制了它在临床诊断中的应用。
肝损伤评估最直观的是病理形态学诊断方法,但手术禁忌症多,只能反应局部的变化,不能反应出肝脏整体的情况。而miRNA-122具有更好的灵敏度和特异性、更小的个体间差异,潜在的分辨致命与非致命性肝损伤的能力以及反映肝损伤模式甚至病因学的能力。因此,miRNA-122具有肝损伤最佳标志物的前景。本研究基于miRFLP的方法对微小核糖核酸122(miRNA-122)进行定量检测。该技术具有很好的稳定性、重复性、准确性和较宽的检测范围,适用于低浓度样本的检测,可用于自动化分析,操作简单,使用方便,各项检测性能均可满足临床检测要求,为临床评估肝损伤提供了一种新的方法,适用于肿瘤患者化疗过程中肝脏损伤的实时监测,以及人体肝脏其他损伤情况的实时检测。该方法应用范围广泛,具有极高的临床应用价值。
| [1] |
Wang K., Zhang S., Weber J., et al. Export of MicroRNAs and MicroRNA-protective Protein by Mammalian Vells[J]. Nucleic Acids Res, 2010, 38(20): 7248-7259. DOI:10.1093/nar/gkq601 |
| [2] |
Mitchell P. S., Parkin R. K., Kroh E. M., et al. Circulating MicroRNAs as Stable Blood-based Markers for Cancer Detection[J]. Proc. Natl. Acad. Sci. U.S.A, 2008, 105(30): 10512-10518. |
| [3] |
Chang J., E Nicolas, D Marks, et al. miR-122, a Mammalian Liver-specific MicroRNA, is Processed from Hcr mRNA and May Downregulate the High Affinity Cationic Amino Acid Transporter CAT-1[J]. RNA Biol, 2004, 1(2): 106-113. DOI:10.4161/rna.1.2.1066 |
| [4] |
Shi Q., Yang X., Mendrick D. L.. Hopes and Challenges in Using miRNAs as Translational Biomarkers for Druginduced Liver Injury[J]. Biomark. Med, 2013, 7(Dili): 307-15. |
| [5] |
US Food and Drug Administration (FDA). FDA Survey to Identify Potential Biomarkers for Qualification[S]. 2015.
|
| [6] |
Wang W., Shi Q., Mattes W. B., et al. Translating Extracellular MicroRNA into Clinical Biomarkers for Drug-induced Toxicity:from High-throughput Profiling to Validation[J]. Biomark. Med, 2015(9): 1177-1188. |
| [7] |
Xie X., Fang T., Zhao Y., et al. MicroRNA-derived Fragment Length Polymorphism Assay[J]. Sci. Rep, 2015, 5(1): 9356. DOI:10.1038/srep09356 |
| [8] |
Zhu B., Zhu W., Ye S., et al. Quantification of MicroRNAs in Human Aqueous Humor by miRFLP Assay[J]. Exp. Eye Res, 2017(162): 73-78. |
| [9] |
NCCLS. EP5-A: Evaluation of Precision Performance of Clinical Chemistry Devices; Approved Guideline[S]. 1992.
|
| [10] |
NCCLS. EP6-A: Evaluation of the Linearity of Quantitative Measurement Procedures: a Statistical Approach Approved Guideline[S]. 2003.
|
| [11] |
NCCLS. EP7-A: Interference Testing in Clinical Chemistry; Approved Guideline[S]. 2002.
|
| [12] |
中国药典: 第四部[S]. 2015: 363-368.
|
| [13] |
Loetscher P., Templer S., Seiverth B., et al. Performance Evaluation of Cobas® HIV-1, a Quantitative Nucleic Acid Test for Use on the Cobas® 6800/8800 Systems[J]. HIV AIDS, 2017, 3(1). DOI:10.16966/2380-5536.135 |
| [14] |
中华医学会肝病学分会药物性肝病学组. 药物性肝损伤诊治指南[J]. 中国病毒病杂志, 2015, 23(5): 1752-1769. |
| [15] |
G. Danan and C. Benichou. Causality Assessment of Adverse Reactions to Drugs-I. A Novel Method Based on the Conclusions of International Consensus Meetings: Application to Drug-induced Liver Injuries[J] J. Clin. Epidemiol., 1993, 46(11): 1323-1330, 1993.
|
| [16] |
Chalasani N. P., Hayashi P. H., Bonkovsky H. L., et al. ACG Clinical Guideline:The Diagnosis and Management of Idiosyncratic Drug-Induced Liver Injury[J]. Am. Gastroenterol J, 2014, 109(7): 950-966. DOI:10.1038/ajg.2014.131 |
| [17] |
P H Hayashi. Causality Assessment in Drug-induced Liver Injury[J]. Seminars in Liver Disease, 2009, 29(4): 348-356. DOI:10.1055/s-0029-1240003 |
| [18] |
SAFE-T. Summary Data Package Novel Clinical Biomarkers of Drug-Induced Liver Injury[EB/OL].[2018-04-20]. http://www.imi-safe-t.eu/.
|
| [19] |
Torres A. G., Fabani M. M., Vigorito E., et al. MicroRNA Fate upon Targeting with Anti-miRNA Oligonucleotides as Revealed by an Improved Northern-blot-based Method for miRNA Detection[J]. Rna, 2011, 17(5): 933-943. DOI:10.1261/rna.2533811 |
| [20] |
É. Várallyay, J. Burgyán, Z. Havelda. MicroRNA Detection by Northern Blotting Using Locked Nucleic Acid Probes[J]. Nat. Protoc., 2008, 3(2): 190-196.
|
| [21] |
Koscianska E., Starega-Roslan J., Sznajder L. J., et al. Northern Blotting Analysis of microRNAs, Their Precursors and RNA Interference Triggers[J]. BMC Mol. Biol, 2011, 12(1): 14. DOI:10.1186/1471-2199-12-14 |
| [22] |
Saito Y., Suzuki H., Taya T., et al. Development of a Novel MicroRNA Promoter Microarray for ChIP-on-chip Assay to Identify Epigenetically Regulated MicroRNAs[J]. Biochem. Biophys. Res. Commu, 2012, 426(1): 33-37. DOI:10.1016/j.bbrc.2012.08.012 |
| [23] |
Kim H., Jin J. Choi, Cho M., et al. A PNA Microarray Platform for miRNA Expression Profiling Using on-chip Labeling Technology[J]. Biochip J, 2012, 6(1): 25-33. DOI:10.1007/s13206-012-6104-9 |
| [24] |
Castoldi M., Benes V., Hentze M. W., et al. miChip:A Microarray Platform for Expression Profiling of MicroRNAs Based on Locked Nucleic Acid (LNA) Oligonucleotide Capture Probes[J]. Methods, 2007, 43(2): 146-152. DOI:10.1016/j.ymeth.2007.04.009 |
| [25] |
Chen C., Ridzon D. A., Broomer A. J., et al. Real-time Quantification of MicroRNAs by Stem-loop RT-PCR[J]. Nucleic Acids Res, 2005, 33(10): 1-9. |
| [26] |
Kroh E. M., Parkin R. K., Mitchell P. S., et al. Analysis of Circulating MicroRNA Biomarkers in Plasma and Serum Using Quantitative Reverse Transcription-PCR (qRT-PCR)[J]. Methods, 2010, 50(4): 298-301. DOI:10.1016/j.ymeth.2010.01.032 |
| [27] |
Pan X., Murashov A. K., Stellwag E. J., et al. Monitoring MicroRNA Expression During Embryonic Stem-cell Differentiation Using Quantitative Real-time PCR (qRTPCR)[J]. Methods Mol Biol, 2010, 650(16): 213-224. |
 2019, Vol. 33
2019, Vol. 33